В предыдущем практикуме я получил филогенетической дерево эволюции доменных архитектур, содержащих домен FAD_binding_1. Данное дерево содержит много клад, но для дальнейшей работы я выбрал одну, включающую в себя белки грибов E3RJ05_PYRTT, C6HQZ6_AJECH, C8VLL8_EMENI, D4AJ76_ARTBC, D5GLK9_TUBMM, E3RJ05_PYRTT, E4UYS4_ARTGP, F0ULY5_AJEC8, G0RR38_HYPJQ.
Последовательности этих белков были скачены из базы данных Uniprot и помещены в файл my_profile.fasta.
Для построения и калибровки профиля я применял пакет HMMER, который установлен на kodomo.
Команда для построения профиля: hmm2build my_profile my_profile.fasta
Команда для калибровки профиля: hmm2calibrate my_profile
Результат работы программы: my_profile
Далее я провел поиск по всем белкам UniProt, содержащих мой домен. Для этого я скачал из базы данных UniProt все полноразмерные последовательности белков в файл domen_pro.fasta и осуществил поиск гомологов.
Примененная команда: hmm2search my_profile domen_pro.fasta > search
Результат работы программы: search
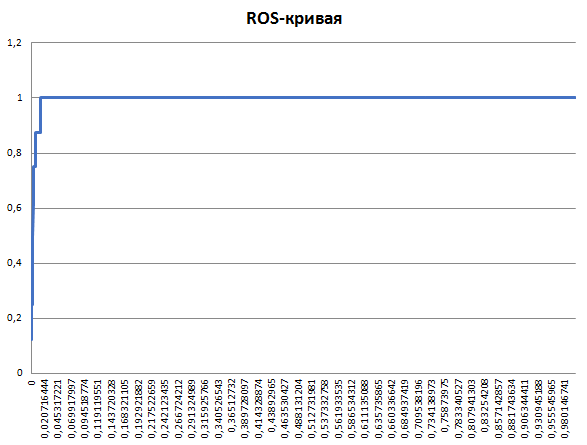
Для анализа результатов я использую ROC-кривую и гистограмму весов находок.
ROC-кривая показывает зависимость количества верно классифицированных положительных примеров от количества неверно классифицированных отрицательных примеров. Она часто используется для анализа качества моделей.
Я перенес результаты поиска в Exsel и отсортировал по убыванию нормализованного веса. Белки-представители моего подсемейства я отметил цифрой 1, остальные цифрой 0. Далее я вычислил показатели чувствительности и специфичности.
По имеющимся данным я построил ROS-кривую.

Легенда графика: по вертикали чувствительность, по горизонтали неспецифичность (1-специфичность)
Теперь необходимо выбрать пороговое значение. Для этого я (в Exsel) рассчитал разность между чувствительностью и неспецифичностью, после чего, используя функцию ПОИСКПОЗ, нашел номер позиции находки, для которой эта разность максимальна (в моем случае это 43).
Так как E-value у таких находок очень мал и в Exsel записался как 0, для сравнения я использовал значения нормализованного веса.
Пороговое значение = 1362
При таком пороге чувствительноть профиля составляет 1,00, а специфичность 0,98575745. Это достаточно хорошие значения, поэтому построенный профиль вполне можно использовать для выделения подсемейства.

Exsel-книга с анализом профиля ROS.xlsx
© Борисов Евгений 2017